空间单细胞DNA测序技术:Topographic Single Cell Sequencing 发表于2018年CELL杂志,文章题目是:Multiclonal Invasion in Breast Tumors Identified by Topographic Single Cell Sequencing31449-6) 非常值得精读。
背景知识
需要仔细查询以下名词的意思:
- Ductal Carcinoma in situ (DCIS)
- Invasive ductal cancer (IDC)
- DCIS-IDC patients
- Topographic Single Cell Sequencing (TSCS)
- the independent lineage model
- the evolutionary bottleneck model
- laser-capture microdissection (LCM)
- whole-genome amplification (WGA)
- degenerative oligonucleotide PCR (DOP-PCR)
- single-nucleus sequencing (SNS)
- Shannon(clonal) diversity
DCIS-IDC的乳腺癌病人的发病区域有3种细胞: normal stromal cells, in situ ducts, invasive cells,后面就是分析着3种细胞的区别。
DCIS病人并不一定会发展成为IDC,这一过程机制不明朗,值得探索。难点在于肿瘤异质性,而且stromal太多,肿瘤细胞太少。
单细胞DNA测序技术很强大,但常规的建库技术都需要细胞悬浮过程,比如 fluorescence-activated cell sorting (FACS),micromanipulation,microdroplets,or nanowells,这样就失去了测序的细胞原来的位置信息,而这个位置信息在研究早期癌症发生进化过程非常重要,所以作者结合了laser-capture microdissection (LCM), laser catapulting, whole-genome amplification (WGA), and single-cell DNA sequencing这4个技术,创造性的开发了Topographic Single Cell Sequencing (TSCS) 技术,并且在10个DCIS-IDC的乳腺癌病人上面应用了,结果表明癌症发生是单一起源,但是在侵袭过程中是多克隆的。技术流程
如下图
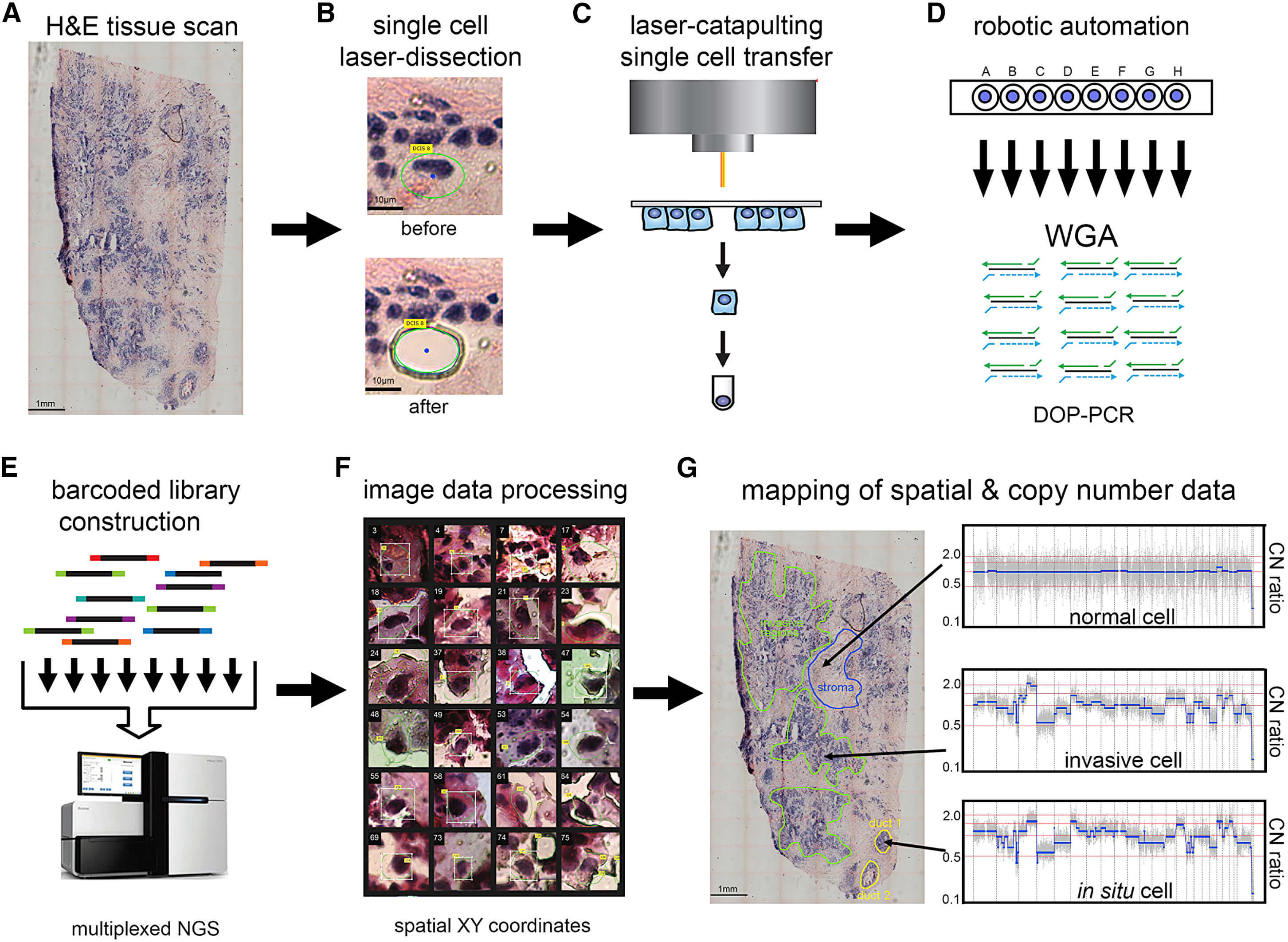
一图胜千言,很容易看明白这个流程,就是先对组织样品进行染色,这样可以区分3种细胞,然后利用LCM技术来挑选微小区域的细胞,再利用laser catapulting精准的挑选一个单细胞去建库测序,单细胞的DNA扩增采取的是DOP-PCR技术,最后进行数据分析,找CNV情况,跟原来的空间位置信息进行关联。8号病人概况
如下图
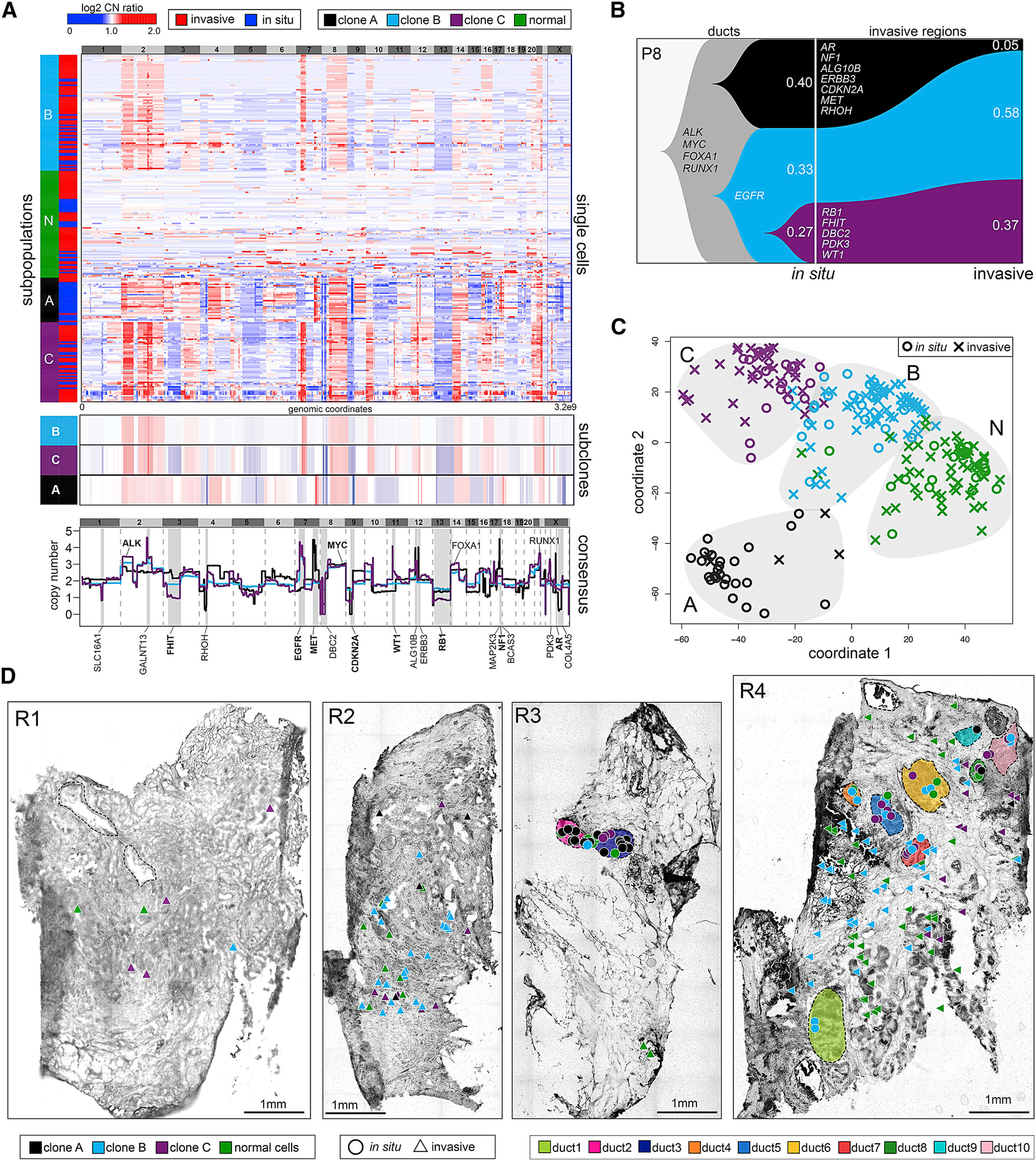
如上图所示,对8号病人,作者从4个病变区域进行采样,总共分析了85 in situ cells and 150 invasive cells,分析测序数据的CNV,使用1-dimensional clustering可以把这些细胞分成4组,其中一组是没有拷贝数变异的二倍体正常细胞。肿瘤细胞分成“A,” “B,” and “C” 这3个亚克隆。使用TimeScape可以分析这些细胞的进化情况。使用multi-dimensional scaling (MDS)分析也得到同样的结果,分成4组,而且可以看到in situ cells 和invasive cells是无法被区分开来的。可以看到3个克隆都起源于ducts,然后A克隆在invasive cells的比例远少于in situ cells ,它的侵袭能力不及B,C克隆。因为B,C克隆部分肿瘤细胞有着EGFR的扩展,这被认为是侵袭能力的象征。4号病人概况
如下图

如上图所示,对4号病人,作者在2额病变区域采用,共分析了46 in situ cells and 58 invasive cells 单细胞DNA测序数据。这个时候分析拷贝数变异得到的是2个肿瘤亚克隆,且都起源于ducts,在侵袭的过程中,B克隆由16% 增长到 67%,但是A克隆由84% 减少到 33%。10个病人整合分析拷贝数变异的进化情况
如下图
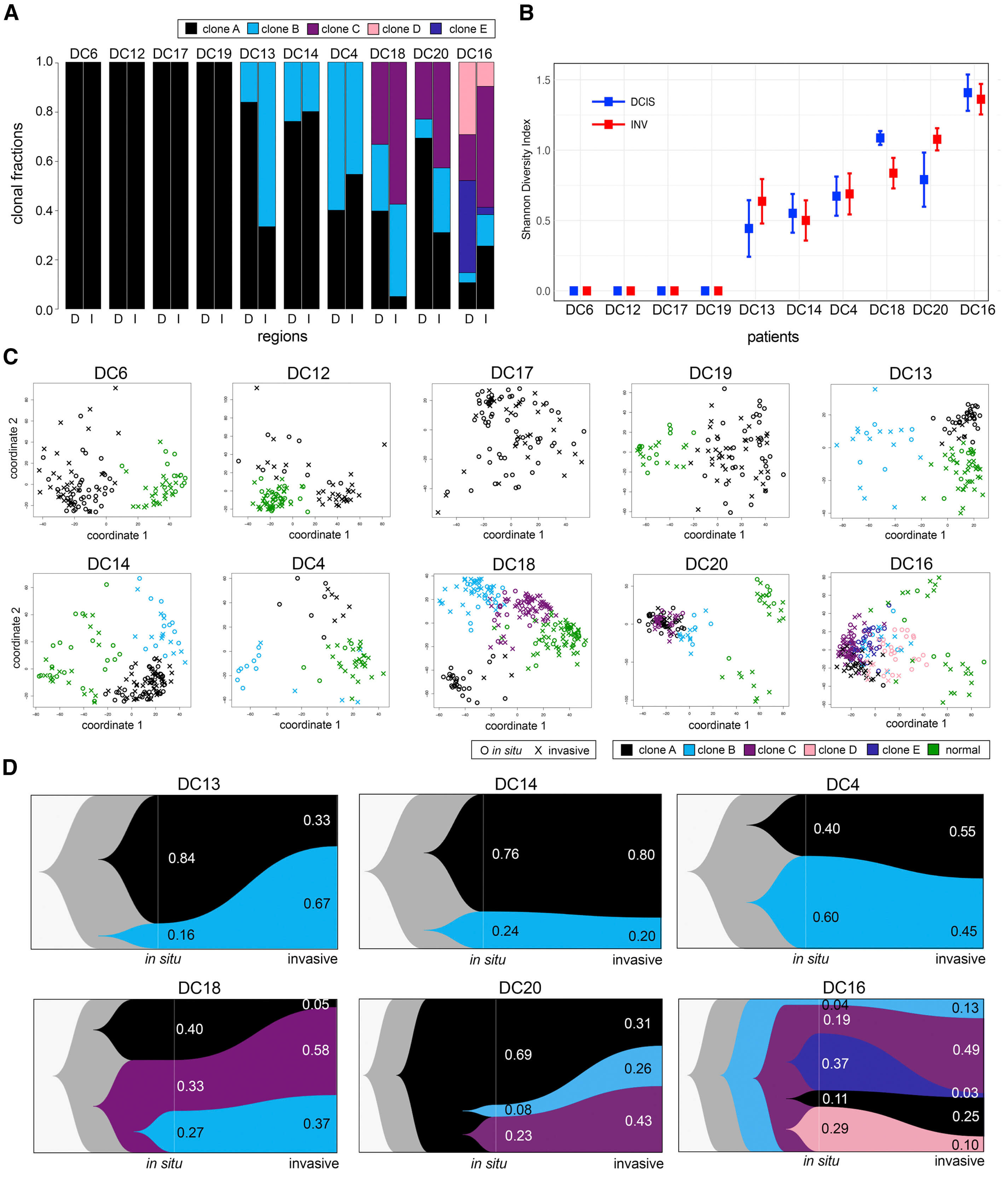
对10个病人综合起来总共测了 425 in situ and 503 invasive 和365 stromal diploid cells,这些单细胞DNA测序数据都是可以从SRA数据库里面下载的。可以看到所有病人的亚克隆都不多,在1~5个之间。比较奇怪的是有4个病人都是单克隆,或者说仅仅是从单细胞DNA测序得到的拷贝数变异无法区分不同的克隆。
即使对于有着多克隆的那些病人来说,并不是所有的克隆在in situ 和 invasive 的比例都发生了变化,说明只有部分克隆是具有高侵袭能力的。肿瘤病人的亚克隆和空间信息的对应
如下图
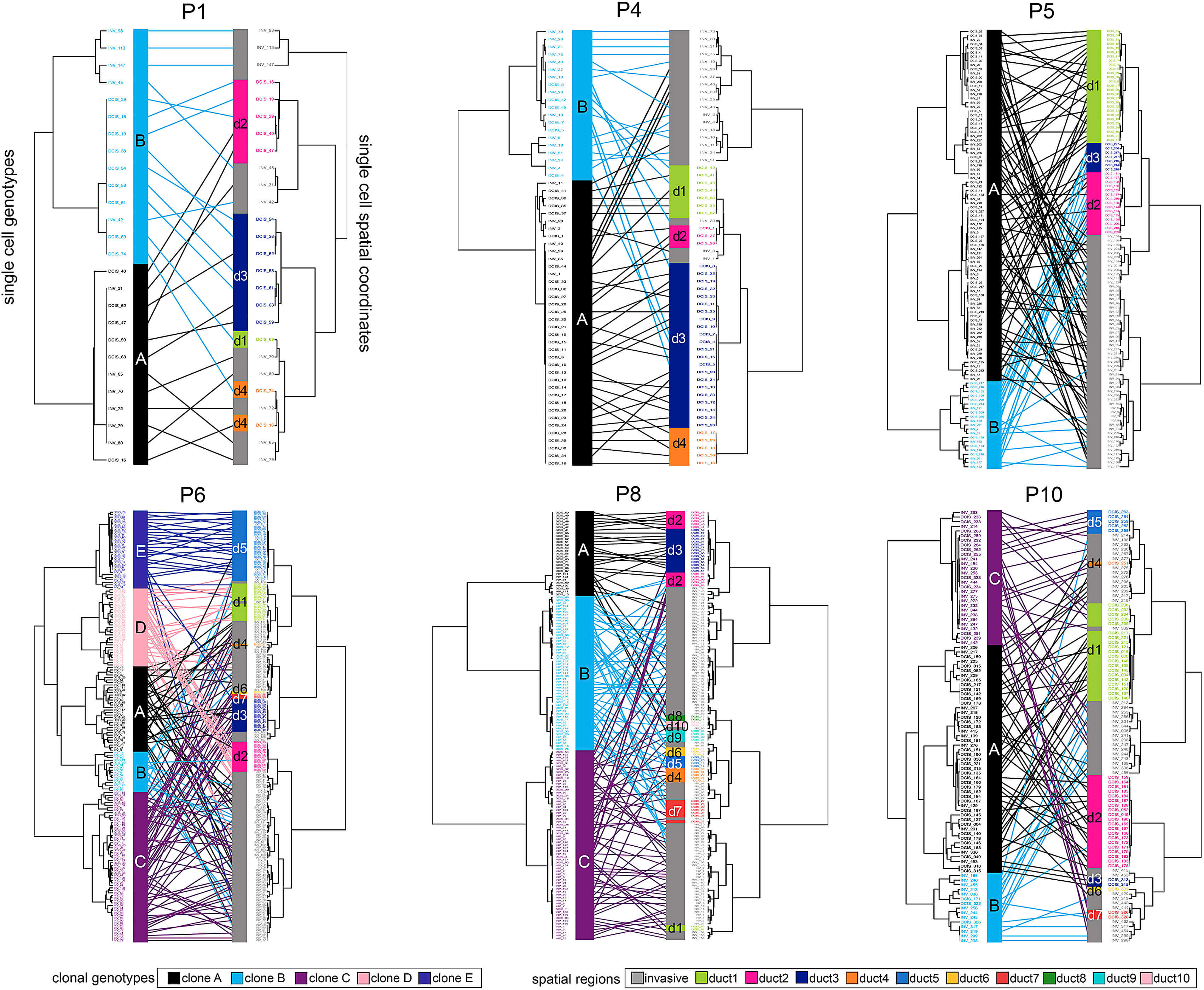
只有6个病人是有着多克隆的,可以看到所以的克隆都会在 in situ and invasive 区域出现,只是不同的克隆在不同的区域的比例不一样,说明这些克隆起源是单一的,但是侵袭是同步的,取决于各自的侵袭能力。3种细胞的外显子测序结果分析
前面背景介绍过DCIS-IDC的乳腺癌病人的发病区域有3种细胞: normal stromal cells, in situ ducts, invasive cells,作者对这10个病人的3种细胞用 laser-capture microdissection (LCM)技术取1000个左右的细胞进行全外显子测序,并且分析somatic mutation情况。
如下图
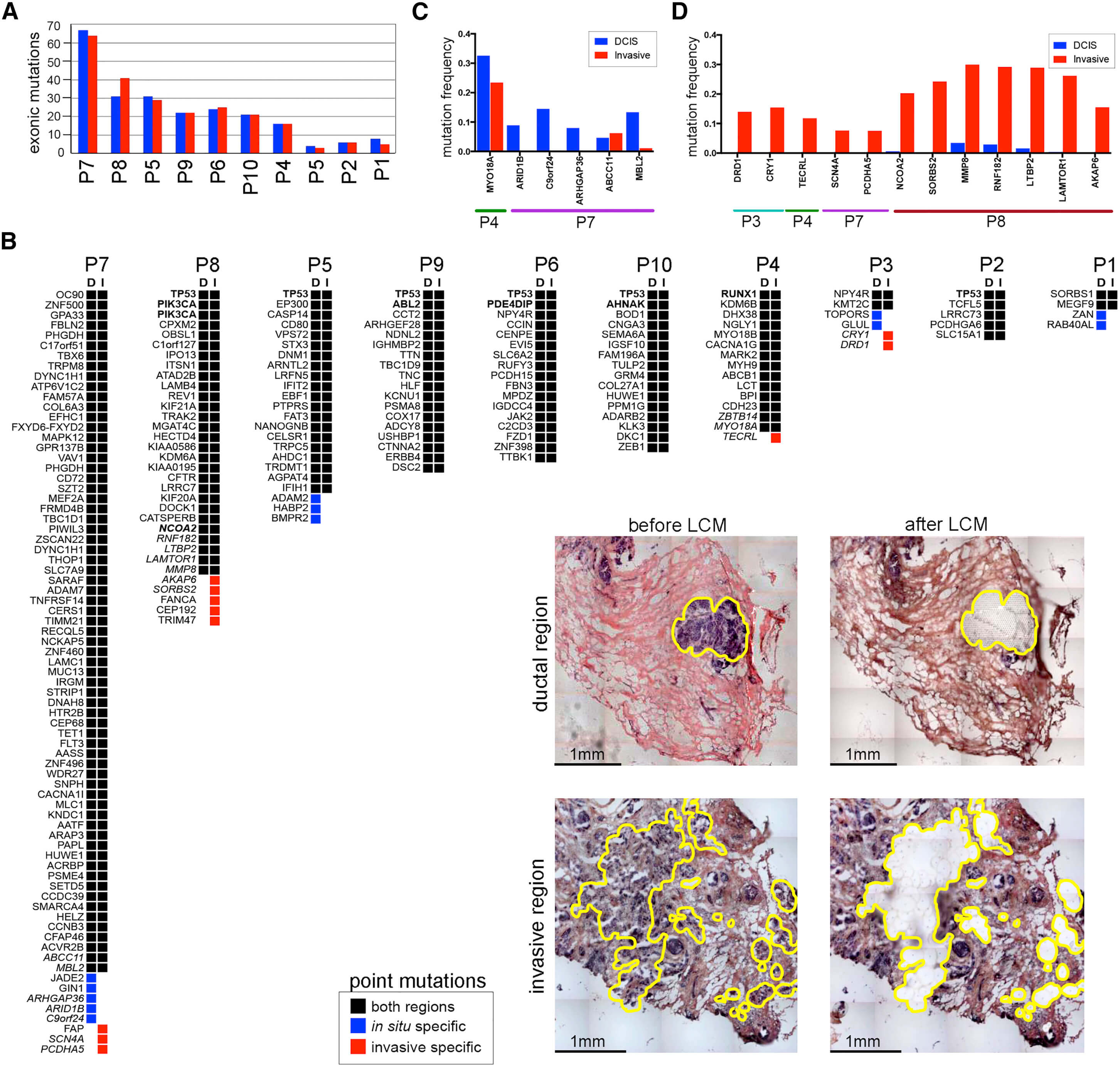
大部分的乳腺癌相关基因,比如TP53, PIK3CA, NCOA2, ABL2, PDE4DIP, AHNAK 都是在ducts和invasive区域都出现了,值得注意的就是 in situ specific (n = 12) or invasive specific (n = 11) in 4 patients (P3, P4, P7, and P8) 这些位点,但是普通的WES技术限制。
所以作者对12个 in situ-specific 的突变进行了45万X的超高深度测序,发现8个的确是真正的 in situ-specific ,另外4个是WES测序技术限制。但是这8个变异并不像是在侵袭过程中起着关键作用,作者并没有解释这是为什么。但是却说另外4个变异里面的MMP8很重要,因为它在ducts的时候频率很低,到了invasion区域就升高了很多,它应该是那些侵袭相关基因。看MAF变化情况
这里作者重点分析 mutation frequency的变化情况
如下图
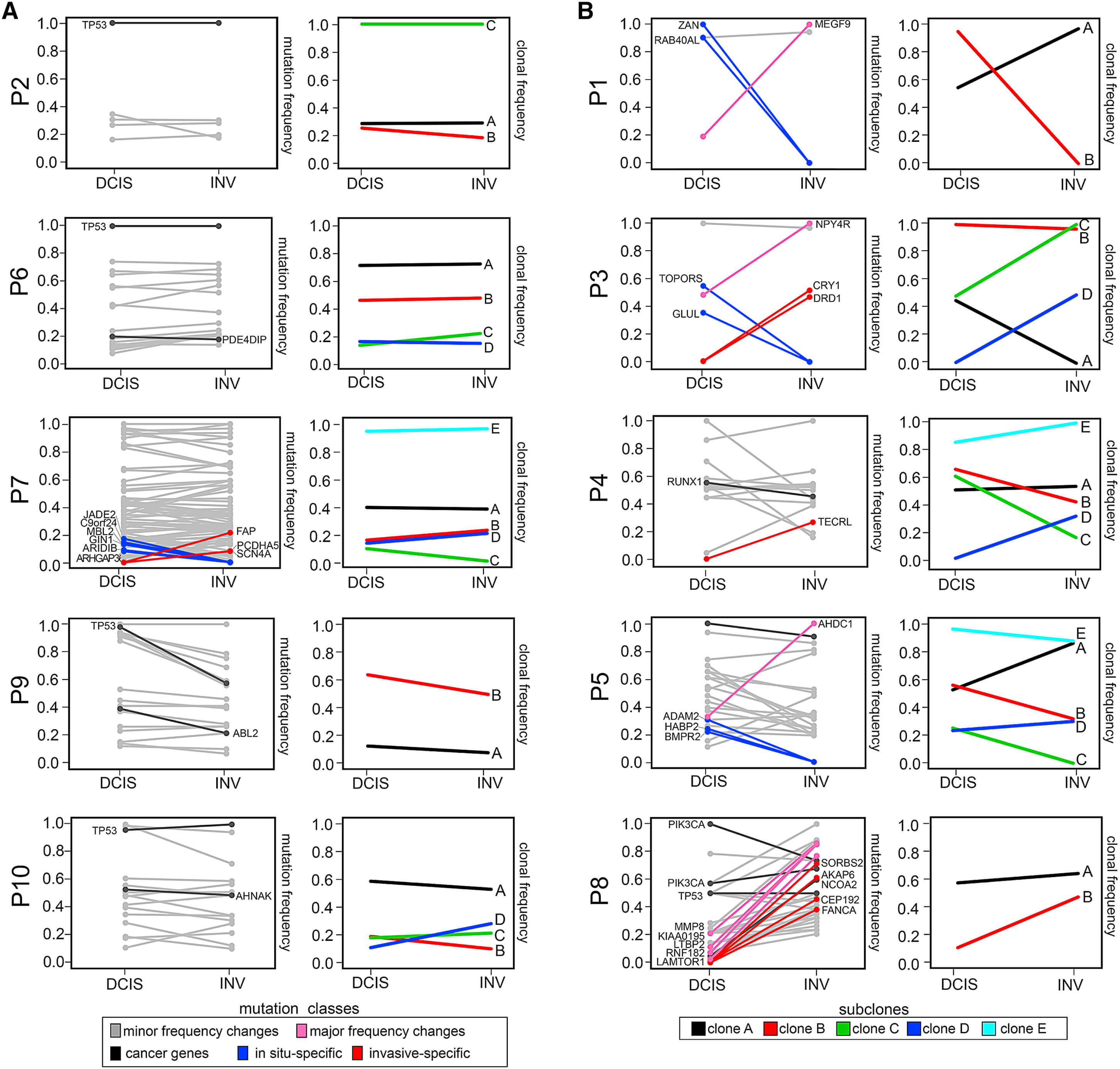
作者就关心那些突变频率发生显著提升的基因,就是 large (>0.5) mutation-frequency changes。的确是发现了7个这样的突变。但是作者并没有解释为什么。
作者接着利用WES数据的somatic mutation信息和CNV信息,使用PyClone 2进行亚克隆推断,发现比前面单细胞的CNV信息推断的要多,作者认为是因为ducts区域的点突变多样性先发生了,还没来得及扩散到invasion区域,所以单细胞的CNV是无法区分的。
还有几个细节我没有仔细介绍,比如TimeScape和PyClone 2的用法,以及作者提出的多克隆侵袭模型。数据公布在SRA
- https://trace.ncbi.nlm.nih.gov/Traces/sra/?study=SRP116771
- https://trace.ddbj.nig.ac.jp/DRASearch/study?acc=SRP116771
其中NCBI的镜像要方便一点,数据量很大:
- https://trace.ddbj.nig.ac.jp/DRASearch/study?acc=SRP116771
- Experiments:1989
- Runs:1989 (1.1Tbp; 472.2Gb)
包含着 10 个病人的 norm,ducts,invasive这3个地方的外显子测序数据(~150X),用来找somatic mutation。
还有从这10个病人里面取的1293个单细胞的DOP-PCR测序,这些数据只用来找CNV了。
还有部分基因的超深度测序(~45万X),看mutation frequency的变化情况,研究超低频突变。
节选部分如下;
| Assay_Type | LibraryLayout | LibrarySelection | Library_Name | MBases |
| ————— | ——————- | ———————— | ———————- | ——— |
| WGA | SINGLE | RANDOM | P10_SC_INV_065 | 136 |
| WGA | SINGLE | RANDOM | P10_SC_INV_066 | 124 |
| WGA | SINGLE | RANDOM | P10_SC_INV_067 | 195 |
| WGA | SINGLE | RANDOM | P10_SC_INV_068 | 458 |
| WXS | PAIRED | Hybrid | P10_Norm_Exome | 25677 |
| WXS | PAIRED | Hybrid | P10_INV_Exome | 12410 |
| WXS | PAIRED | Hybrid | P10_DCIS_Exome | 45593 |
| WGA | SINGLE | RANDOM | P10_SC_DCIS_076 | 358 |
| WGA | SINGLE | RANDOM | P10_SC_DCIS_077 | 272 |
| WGA | SINGLE | RANDOM | P10_SC_DCIS_078 | 341 |
| WGA | SINGLE | RANDOM | P10_SC_DCIS_079 | 340 |
| WGA | SINGLE | RANDOM | P10_SC_DCIS_080 | 323 |
- Runs:1989 (1.1Tbp; 472.2Gb)
323