可变剪切是指mRNA前体以多种方式将exon连接在一起的过程。 由于可变剪切使一个基因产生多个mRNA转录本,不同mRNA可能翻译成不同蛋白。
可变剪切背景知识
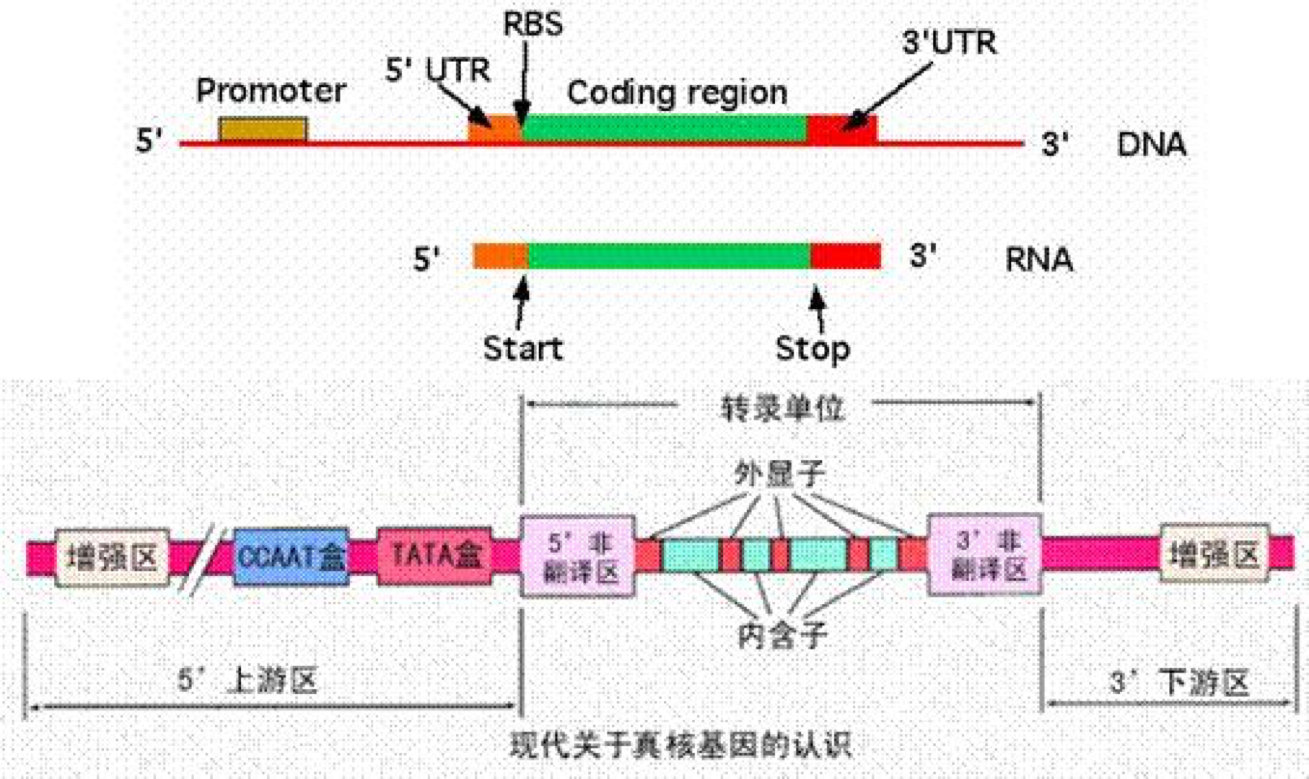
转录组一般是指从细胞或组织的基因组所转录出来的RNA的总和,包括编码蛋白质的mRNA和各种非编码RNA(rRNA,tRNA,snRNA,snoRNA,lncRNA,microRNA等)。真核生物的基因结构是不连续的,如下图:
其基因组最初的转录产物其实并不是成熟的mRNA分子,而是它的前体pre-mRNA,那么怎么变成成熟的mRNA呢,就需要从pre-mRNA中将非编码蛋白质的内含子(intron)切除,然后拼接剩下的编码蛋白质的外显子(exon)。但实际上,在这个过程中,有多种多样的前切和拼接方式,从而产生不同的剪切异构体,也就咱们要说的可变剪切。
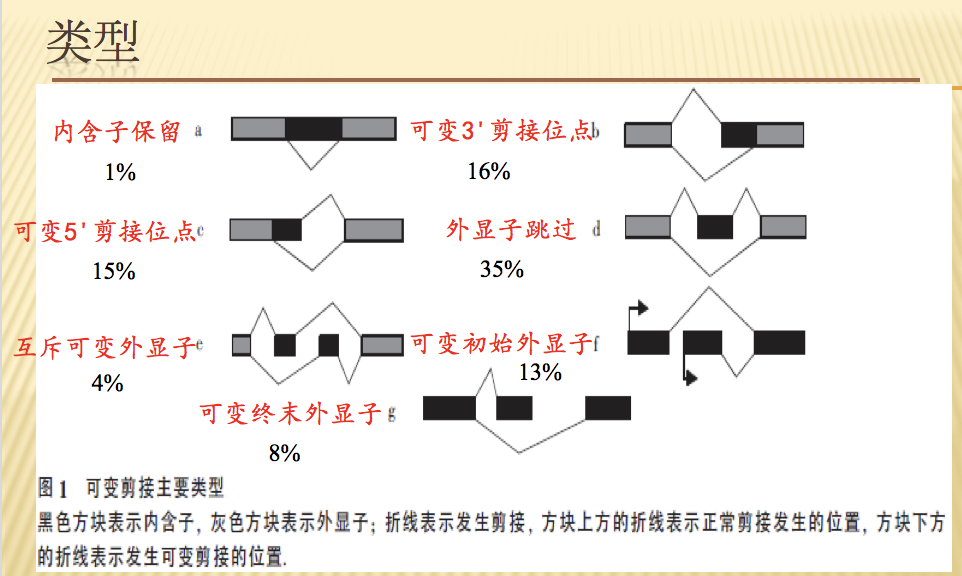
可变剪切的形式复杂多样,大致可以分为5大类。
- 第一类是外显子跳跃型(exon skipping),发生跳跃的外显子和其两侧的内含子都被剪切掉,上游和下游的外显子被直接连着一起保留在剪切后的产物中。
- 第二类是内含子滞留型(intron retention),某一段核苷酸序列在一个剪切体中是外显子的一部分,而在与之对照的剪切体中却是内含子而被剪切掉。
- 第三类是可变5’或3’端剪切(alternative 5’ss splice or alternative 3’ss splice,其中5’ss称供体位点,3’ss称受体位点),和与它对照的另一个剪切体相比,发生剪切的位点在5’或3’端不同,除此,其他剪切选择一致。
- 第四类是转录起始区域可变剪切(alternative TSS),发生剪切的位点在转录起始区域,即与之对应的另一个剪切体除转录起始位点不同外,其余一致。
- 第五类是转录终止区域可变剪切(alternative TTS),与第四类对应,发生剪切的位点只是在转录终止位点不同。
软件算法
比较旧的分析可变剪切的软件主要有SpliceR、SpliceGrapher、ASprofile以及Splicing Express等,它们是基于cufflinks软件的结果,将reads回帖到基因组序列后,根据位置和长度及结构信息,来确定或预测可能的剪切体的类型。目前主流已经不再使用tophat+cufflinks流程了。
比较新的几款软件: