实在是不能理解这么出名的软件居然会有如此明显的错误,让我感到很伤心,homer听过的人都知道它是用来做peaks注释的,其实本质上就是把自己找到的genomic region注释到一致的genomic region(intron,exon,promoter,tss,tts,utr)上面。
这种注释必然是批量的,大部分情况的注释也绝对是正确的,不然这个错误怎么会现在才被我发现呢
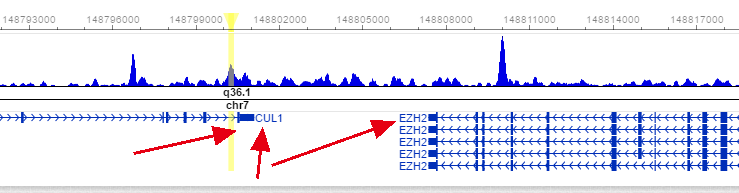
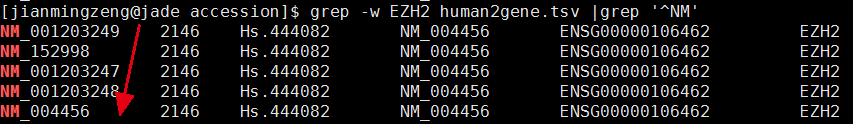
peak14938 chr7 148800534 148800697 + 6.44164 NA 3' UTR (NM_003592, exon 22 of 22) 3' UTR (NM_003592, exon 22 of 22) 82894 NM_001203249 2146Hs.444082 NM_004456 ENSG00000106462 EZH2
可以看到,它把这个chr7 148800534 148800697区域注释到了3' UTR (NM_003592, exon 22 of 22)这个没有错,但是悲催的是后面它给映射到了EZH2基因
我不明白它是如何犯下这个严重的错误的:
很明显,EZH2基因的refseq ID里面并没有NM_003592,NM_003592这个ID是属于CUL1基因的。
我总不能去看源码,找找它的错误出在哪里吧!
但是有了这个错误,这个前车之鉴,我如何能相信它其它的结果呢?
后来我仔细看了它的readme,发现不是它错了,而是我理解错了:http://homer.ucsd.edu/homer/ngs/annotation.html
它注释的列如下:
- Peak ID
- Chromosome
- Peak start position
- Peak end position
- Strand
- Peak Score
- FDR/Peak Focus Ratio/Region Size
- Annotation (i.e. Exon, Intron, ...)
- Detailed Annotation (Exon, Intron etc. + CpG Islands, repeats, etc.)
- Distance to nearest RefSeq TSS
- Nearest TSS: Native ID of annotation file
- Nearest TSS: Entrez Gene ID
- Nearest TSS: Unigene ID
- Nearest TSS: RefSeq ID
- Nearest TSS: Ensembl ID
- Nearest TSS: Gene Symbol
- Nearest TSS: Gene Aliases
- Nearest TSS: Gene description
- Additional columns depend on options selected when running the program.
其中annotation那一列,并非一定要与Nearest TSS:的基因一致!!!!
因为有些基因的是尾巴接着尾巴,这样即使某个region的确定位在A基因的尾巴上面,但是它距离A基因的TSS要比B基因的TSS远!!!
所以就出现了基因不一致的情况。