想了想还是写一个系列教程吧,问的朋友也太多了,主要是因为cytoscape跟python一样,经历了从2到3的进化阵痛过程,而且进化的面目全非了!!!很多人拿着2.x的说明书教程,视频,然后下载的却是3.x版本的cytoscape,真可怕!!!
已经从两万个芯片探测到的基因里面找到了近千个差异基因了,对它们做了GO/KEGG分析还是抓不住重点,看到文献说可以用PPI数据库做network analysis之后找hub基因,也也许可以说明一些问题!
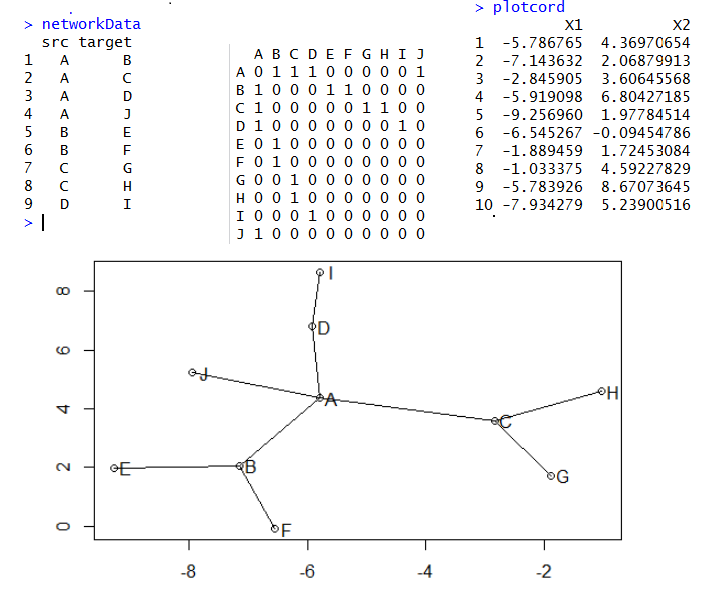
提到 network analysis ,我想起来我以前总结过 R语言画网络图的三部曲,里面讲到过网络分析的基本原理!
网络图是为了展示数据与数据之间的联系,在生物信息学领域,一般是基因直接的相互作用关系,或者通路之间的联系!
通俗点说,就是我有一些点,它们之间并不是两两相互联系,而是有部分是有连接的,那么我应该如何把这些点画在图片上面呢?因为这些都并没有X,Y坐标,只有连接关系,所以我们要根据一个理论来给它们分配坐标,这样就可以把它们画出来了,然后也可以对这些点进行连线,连完线后,就是网络图啦!!!
而给它们分配坐标的理论有点复杂,大概类似于物理里面的万有引力和洛仑磁力相结合来给它们分配一个位置,使得总体的能量最小,就是最稳定状态!而通常这个状态是逼近,而不是精确,所以我们其实对同样的数据可以画出无数个网络图,只需使得网络图合理即可!

看这个图,就明白了,网络图,就是在画布上给感兴趣的点一个坐标,通过统计方法,有两个:Two of the most prominente algorithms (Fruchterman & Reingold’s force-directed placement algorithm and Kamada-Kawai’s)