其实国际三大主流生物信息学数据库运营单位都出了自己的基因组坐标转换,它们分别是 (UCSC liftOver, NCBI Remap, Ensembl API)
Ensembl's Assembly Converter.是基于crossmap的,我觉得挺好用的,就介绍给大家!!!
This online tool currently uses CrossMap, which supports a limited number of formats (see our online documentation for details of the individual data formats listed below). CrossMap also discards metadata in files, so track definitions, etc, will be lost on conversion.
Important note: CrossMap converts WIG files to BedGraph internally for efficiency, and also outputs them in BedGraph format.
但是不知道为什么UCSC的liftover最出名,我也写过它的教程,(http://www.bio-info-trainee.com/?p=990)
还算好用,比较真正基因组坐标转换的需求很少,但是略有一些限制,所以我用起来了Ensembl的crossmap软件。
该软件说明书就在主页,很简单的:http://crossmap.sourceforge.net/
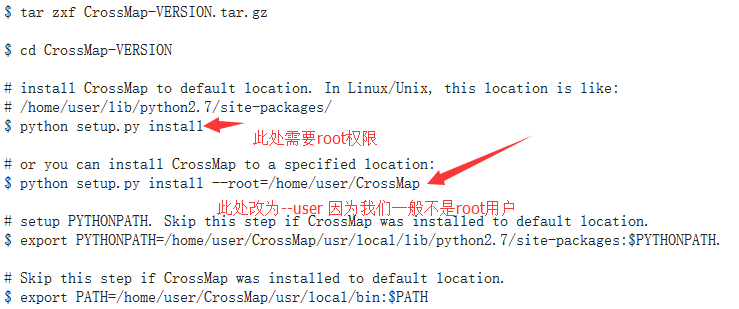
一、程序安装
直接wget到linux系统即可,解压之后就能看到该程序,是基于Python的,所以安装方式跟Python模块一样!
软件官网也有安装说明书
我的安装命令是:
python setup.py install --user
安装之后添加到环境变量
# setup PYTHONPATH. Skip this step if CrossMap was installed to default location.
$ export PYTHONPATH=/home/jmzeng/.local/lib/python2.7/site-packages:$PYTHONPATH.
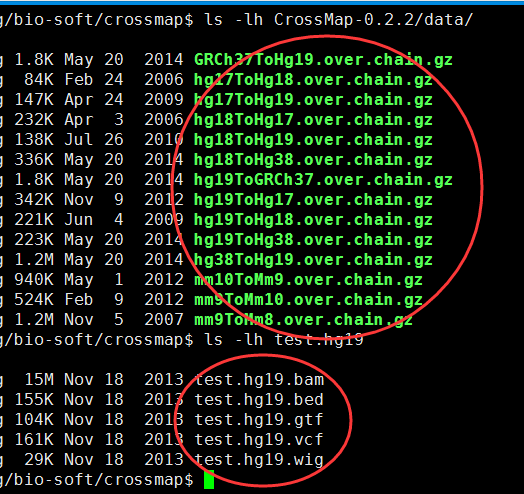
二、输入数据准备
该软件提供了测试数据,也提供了基因组转换的模板chain格式文件:
它要求的待转换文件很广泛:
三、运行程序
找到你的crossmap安装目录,根据你的安装命令来的!
我的程序在/home/jmzeng/.local/bin 里面,直接可以使用
根据下面的例子,自己修改就可以了,程序用全路径调用,然后写明是什么文件格式,然后列出chain文件的地址,然后input和output即可
Example (run CrossMap with no output_file specified):
$ python CrossMap.py bed hg18ToHg19.over.chain.gz test.hg18.bed3
Conversion results were printed to screen directly (column1-3 are hg18 based, column5-7 are hg19 based):
chr1 142614848 142617697 -> chr1 143903503 143906352
chr1 142617697 142623312 -> chr1 143906355 143911970
chr1 142623313 142623350 -> chr1 143911971 143912008
chr1 142623351 142626523 -> chr1 143912009 143915181
chr1 142633862 142633883 -> chr1 143922520 143922541
chr1 142633884 142636152 -> chr1 143922542 143924810
chr1 142636152 142636326 -> chr1 143924813 143924987
chr1 142636339 142636391 -> chr1 143925000 143925052
chr1 142636392 142637362 -> chr1 143925052 143926022
chr1 142637373 142639738 -> chr1 143926033 143928398
chr1 142639739 142639760 -> chr1 143928399 143928420
chr1 142639761 142640145 -> chr1 143928421 143928805
chr1 142640153 142641149 -> chr1 143928813 143929809
四、输出数据解读
输出数据没什么好解读的了,进去的是什么数据,出来的就是什么数据,只是把你的坐标进行了转换
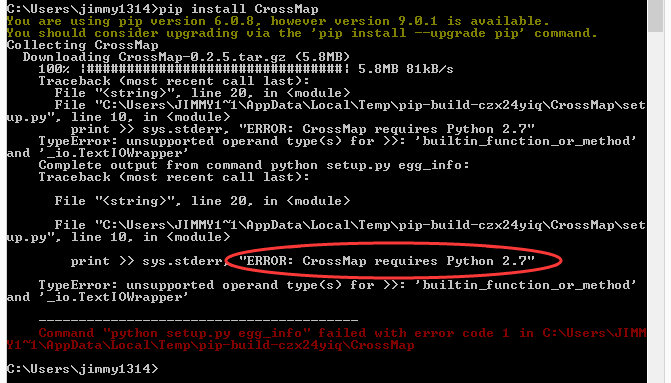
有个比较重要的事情,这个程序需要python2.7 其它版本不友好!