作用:可以检测别人是否把自己的样本搞混,也可以看看测序是否分布均匀!
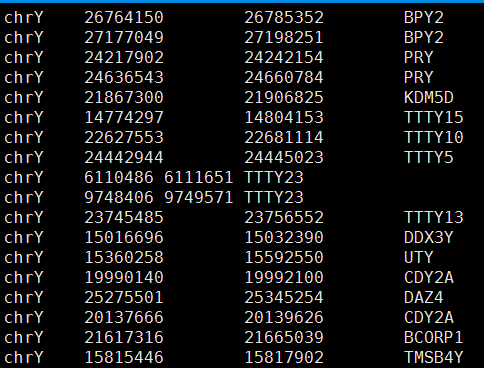
首先,我们要拿到Y染色体上面的基因的坐标信息!
因为我们的是hg19,所以我们要下载hg19的基因信息!
我们首先解析refGene文件,找到chrY的unique基因!
这四列分别是:chromosome/start/end/gene_symbol
4程序如下:
[perl]
open FH,"/home/jmzeng/hg19/chrY.gene.special.position" or die "file error !!!";
while(<FH>){
chomp;
@F=split;
foreach ($F[1]..$F[2]){
$h{$_}=$F[3];
}
$length{$F[3]}=$F[2]-$F[1]+1;
}
close FH;
open FH,$ARGV[0];
while(<FH>){
chomp;
@F=split;
next unless $F[0] eq 'chrY';
next if $F[2]<20;
if (exists $h{$F[1]}){
$count{$h{$F[1]}}++ ;
}else{
$count{'other'}++ ;
}
}
close FH;
print "$_\t$length{$_}\t$count{$_}\n" foreach sort keys %count;</pre>
</div>
<div>[/perl]
对一个男性样本,结果会如下:
gene/length/pos
| AMELY | 8111 | 1269 |
| BCORP1 | 47724 | 689 |
| CSPG4P1Y | 3799 | 538 |
| DAZ1 | 69739 | 762 |
| DAZ2 | 71901 | 228 |
| DAZ3 | 73222 | 233 |
| DAZ4 | 73222 | 540 |
| DDX3Y | 12825 | 3654 |
| EIF1AY | 17445 | 929 |
| FAM224A | 4295 | 82 |
| FAM224B | 4293 | 85 |
| GOLGA2P3Y | 4866 | 68 |
| GYG2P1 | 15476 | 547 |
| HSFY2 | 42277 | 3950 |
| KDM5D | 39526 | 7425 |
| NLGN4Y | 319396 | 3872 |
| PCDH11Y | 105374 | 6627 |
| PRKY | 107577 | 1390 |
| PRORY | 3388 | 735 |
| RBMY1B | 14451 | 232 |
| RBMY1D | 14411 | 117 |
| RBMY1E | 14410 | 157 |
| RBMY1J | 14407 | 65 |
| RBMY2EP | 6416 | 27 |
| RBMY2FP | 7348 | 419 |
| RPS4Y1 | 25376 | 1856 |
| RPS4Y2 | 24966 | 1831 |
| SRY | 888 | 703 |
| TBL1Y | 180999 | 3231 |
| TGIF2LY | 958 | 808 |
| TMSB4Y | 2457 | 534 |
| TSPY4 | 132211 | 1525 |
| TTTY14 | 205048 | 394 |
| TTTY4C | 36811 | 39 |
| TTTY9A | 9317 | 580 |
| TXLNGY | 23067 | 1968 |
| USP9Y | 159610 | 10508 |
| UTY | 232293 | 6670 |
| VCY | 742 | 291 |
| XKRY2 | 1582 | 980 |
| ZFY | 47437 | 3125 |
| other | 100328 |
对女性样本,结果会如下;
| NLGN4Y | 319396 | 575 |
| PCDH11Y | 105374 | 1643 |
| PRKY | 107577 | 82 |
| TGIF2LY | 958 | 191 |
| TTTY14 | 205048 | 139 |
| other | 54297 |
从结果可以看出来,很多基因都是y染色体特有的,这个结果是表明我们的测序非常棒