针对高通量测序数据,包括WGS,WES,甚至snp6芯片也行。
我这里主要讲根据vcf文件里面的chrX的纯合率来判断性别,大家可以自己处理数据得到vcf文件!
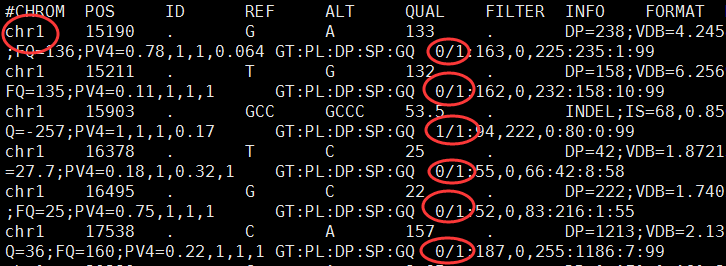
主要是读取vcf文件,选择chrX上面的记录,统计genotype即可:
我这里拿之前的自闭症项目数据来举例子:
根据数据提供者的信息,3-4-5分别就是孩子、父亲、母亲,统计chrX的snp的的纯合和杂合的比例,代码很简单
vcf文件一般第一列是染色体,第6列是质量,第10列是基因型已经测序深度相关信息
cat Sample5.gatk.UG.vcf |perl -alne '{next unless $F[0] eq "chrX";next unless $F[5]>30;$h{(split/:/,$F[9])[0]}++}END{print "$_\t$h{$_}" foreach keys %h}'
如果纯合的snp是杂合的倍数超过一定阈值,比如4倍,就可以判断是男性。
对于Sample3来说,很明显,是男孩,因为X染色体都是纯合突变
0/1 391
1/1 2463
2/2 1
1/2 32
0/2 1
对于Sample4来说,很明显,应该是母亲,证明之前别人给我的信息有误
1/1 3559
1/2 27
0/1 1835
0/2 5
那么Sample5很明显就是父亲咯
1/1 2626
0/1 356
1/2 22
1/1 2463
2/2 1
1/2 32
0/2 1
对于Sample4来说,很明显,应该是母亲,证明之前别人给我的信息有误
1/1 3559
1/2 27
0/1 1835
0/2 5
那么Sample5很明显就是父亲咯
1/1 2626
0/1 356
1/2 22