NCBI的taxid简单介绍
物种的信息集合都在它的NCBI的taxid号里面,在NCBI里面关于它的英文介绍地址如下 http://www.ncbi.nlm.nih.gov/guide/taxonomy/ ,NCBI人为的给自然界所有的物种都给了一个编号,这个编号就是taxid,是根据计算机里面树这种数据结构来编码的,其中人类的编号是 9606,7227是果蝇,我们只需要进入这个物种的taxid里面就能看的关于它的一切NCBI存在并且收集好的信息。
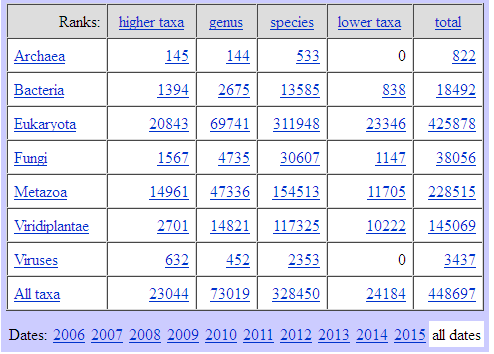
可以看到NCBI到2015年为止已经收录近50万的物种的基因。
我们可以进入人类的9606这个ID里面进去看看
http://www.ncbi.nlm.nih.gov/taxonomy/?term=9606[uid]
可以看出,人类这个并不是最底层的taxid类别,下面还有两个分类
关于人类这个物种的信息是非常多的
但是它下面的两个亚种人,就比较少的信息。
关于这个taxid的资料还有很多 ftp://ftp.ncbi.nlm.nih.gov/pub/taxonomy/
其中我们可以下载 wget ftp://ftp.ncbi.nih.gov/pub/taxonomy/taxdump.tar.gz 这个文件,解压可以看到里面有以下文件,其中比较重要的两个文件
nodes.dmp 这个文件列出了taxid的树的结构信息,子节点,父节点等等
names.dmp 这个文件里面列出了每个独特的taxid对应着的物种名
其它信息不重要,自己看咯
gencode.dmp
-----------
Genetic codes file:
genetic code id -- GenBank genetic code id
abbreviation -- genetic code name abbreviation
name -- genetic code name
cde -- translation table for this genetic code
starts -- start codons for this genetic code
delnodes.dmp
------------
Deleted nodes (nodes that existed but were deleted) file field:
tax_id -- deleted node id
merged.dmp
----------
Merged nodes file fields:
old_tax_id -- id of nodes which has been merged
new_tax_id -- id of nodes which is result of merging
citations.dmp
-------------
Citations file fields:
cit_id -- the unique id of citation
cit_key -- citation key
pubmed_id -- unique id in PubMed database (0 if not in PubMed)
medline_id -- unique id in MedLine database (0 if not in MedLine)
url -- URL associated with citation
text -- any text (usually article name and authors)
-- The following characters are escaped in this text by a backslash:
-- newline (appear as "\n"),
-- tab character ("\t"),
-- double quotes ('\"'),
-- backslash character ("\\").
taxid_list -- list of node ids separated by a single space